So entsorgen Zellen ihre überschüssigen Genprodukte
Zelleigene Entsorgung: Ein internationales Team um Sigurd Braun (bis vor kurzem Arbeitsgruppenleiter am BMC, Lehrstuhl Physiologische Chemie) hat den Abbau von überzähligen Ribonukleinsäure-Molekülen (RNA) im Zellkern untersucht. Diese Studie war Teil des Sonderforschungsbereichs 1064 „Chromatin Dynamik“ und wurde im September im Fachjournal Nature Structural & Molecular Biology veröffentlicht. Sigurd Braun ist kürzlich im Rahmen einer Heisenberg-Professur an die Justus-Liebig-Universität Gießen gezogen.
Die Genexpression steuert die Neusynthese von Proteinen, die bestimme Aufgaben in der Zelle wahrnehmen. Dies geschieht durch das Ablesen von Genen und Anfertigung von Kopien in Form von sogenannten Boten-RNAs (messenger-RNA, mRNA). Dieser Prozess wird als Transkription bezeichnet. Anschließend werden die mRNA-Moleküle aus dem Zellkern transportiert und in Proteine übersetzt (Translation). Vielzellige Organismen wie der Mensch verfügen über eine große Anzahl unterschiedlicher Zellen, die jeweils eine andere Ausstattung von Proteinen haben. Daher muss die Bereitstellung der mRNAs angepasst werden.
Eine Möglichkeit, die Menge an mRNAs zu regulieren, ist über deren gezielten Abbau. Im Zellkern geschieht dies über eine Abbaumaschinerie, die als nukleäres Exosom bezeichnet wird und auch andere, sog. nicht-kodierende RNAs (ncRNA) abbaut. Um den Abbau dieses breiten Spektrums an RNAs zu bewerkstelligen, arbeitet das Exosom mit Adapter-Faktoren zusammen, die die Erkennung und Weiterleitung der verschiedenen RNA-Moleküle vermitteln. Wo aber genau dieser Abbau im Zellkern stattfindet und wie dieser koordiniert wird, war bislang wenig verstanden.
Zusammen mit internationalen Partnern konnte das Team von Sigurd Braun zeigen, dass verschiedene Routen für den RNA-Abbau im Zellkern existieren. Einer dieser Abbauwege verläuft über ein Protein der Kernmembran, die den Zellkern umschließt. Dieses Protein, Lem2, kommt in allen Eukaryonten von der einzelligen Hefezelle bis zum Menschen vor.
Zellen, denen Lem2 fehlt, akkumulieren ncRNA und mRNA, die normalerweise vom Exosom abgebaut werden. „Dies war für uns der erste Hinweis, dass der RNA-Abbauweg defekt ist,“ erklärt Sigurd Braun. Bei der Aufklärung des molekularen Mechanismus fanden die Forschenden dann heraus, dass Lem2 tatsächlich Teil des Exosom-Abbauwegs ist und direkt an einen der Adapter-Faktoren bindet. Weiter fanden sie heraus, dass RNA-Moleküle, die über das Exosom abgebaut werden, oft an der Kernmembran lokalisiert sind. Umgekehrt werden diese RNAs aber von den Adapter-Faktoren nicht richtig erkannt, wenn Lem2 fehlt. Dies alles deutet darauf hin, dass Lem2 eine zentrale Stellung in diesem Abbauweg einnimmt und das Zusammenspiel zwischen RNA-Erkennung und Weiterleitung zum Exosom an der Kernmembran koordiniert.
Warum ist es aber nun wichtig, die Abbaumaschinerie an der Kernmembran zu verankern? „Durch die Rekrutierung an die Kernmembran kommt es zu einer Konzentrierung der beteiligten Faktoren, die andernfalls verteilt im Zellkern vorliegen würden und sich vielleicht nicht finden würden,“ sagt Sigurd Braun. „Auch hilft dies vermutlich, die einzelnen Schritte von der Erkennung der RNAs bis zu deren Abbau besser zu koordinieren“.
Interessanterweise ist Lem2 auch noch an weiteren Aufgaben im Zellkern beteiligt, wie z.B. dem Abschalten von Retrotansposons, die auch als „springende Gene“ bekannt sind und die die Integrität des Genoms gefährden. Wie Lem2 diese unterschiedlichen Aufgaben an der Kernmembran ausübt, bleibt eine spannende Frage, die das Forscherteam um Sigurd Braun in den nächsten Jahren beantworten möchte.
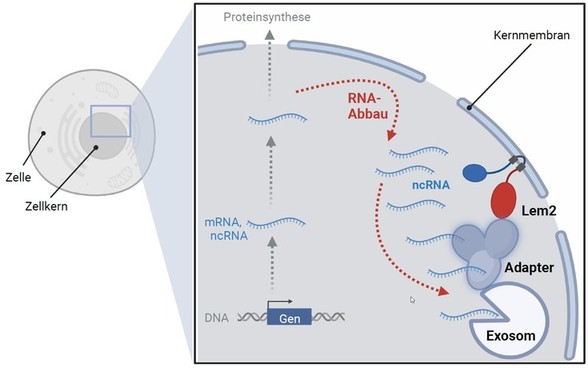
Modell der Funktion von Lem2 im RNA-Abbau
Publikation: The inner nuclear membrane protein Lem2 coordinates RNA degradation at the nuclear periphery. L. Martín Caballero, M. Capella, R.R. Barrales et al. Nat Struct Mol Biol 29, 910–921 (2022).

